Génétique quantitative des normes de réactions: une partition en oignon
OK, bon… ce projet remonte à… 2018 ?! Lorsque j’ai commencé mon post-doctorat avec Luis-Miguel Chevin, on a discuté de la partition de la variance phénotypique dans un contexte de plasticité phénotypique. En particulier, on a discuté de certaines limites qu’on constatait dans un article de méta-analyse publié en 2014. Nous voulions concevoir une partition de variance qui puisse tenir compte de la contribution de la « forme » des normes de réaction à la variance phénotypique totale. Il nous a donc fallu 7 ans pour publier tout ça et près d’un an de plus pour que j’en fasse un billet sur ce site…

Mais pourquoi partitionner la variance de la plasticité phénotypique ?
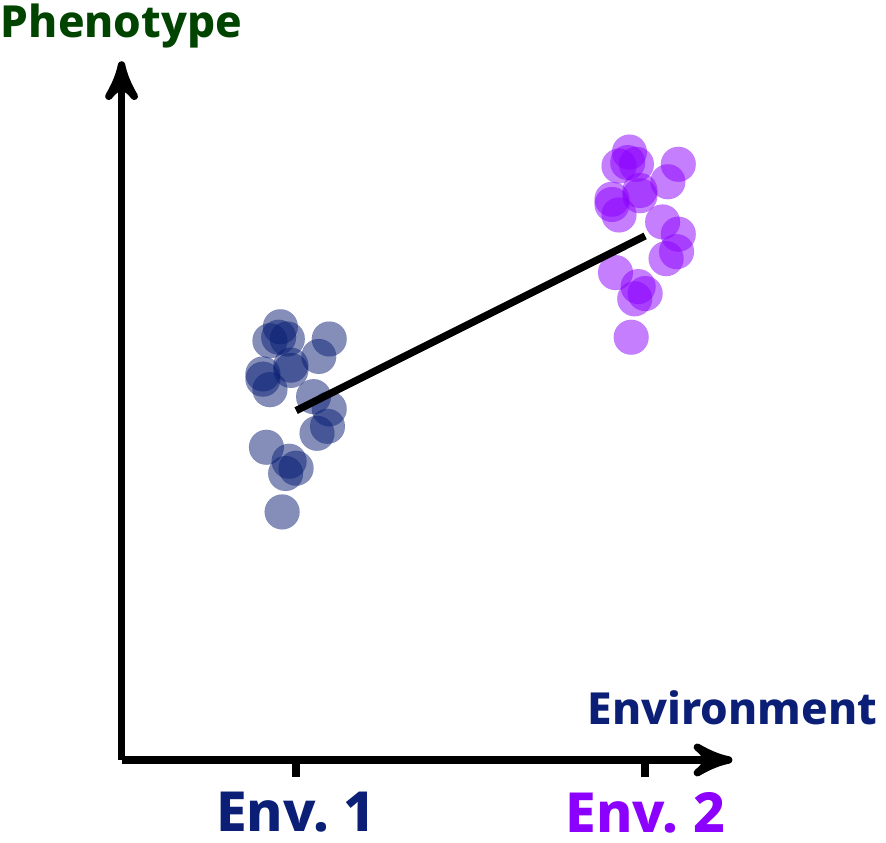
Les traits phénotypiques sont souvent plastiques, ce qui signifie que leur expression dépend de l’environnement dans lequel ils s’expriment. Une façon d’étudier cette plasticité phénotypique passe par l’utilisation de normes de réaction : la prédiction de la valeur phénotypique moyenne qu’un trait prendra dans un environnement donné, pour un génotype donné. Mathématiquement, on peut définir cette norme de réaction comme une fonction qui prend en compte les valeurs environnementales et génotypiques et produit une valeur phénotypique, comme le fait la ligne noire dans le graphique à gauche.
Mais la forme que prend cette fonction n’est pas nécessairement une ligne droite (bien que ce soit une hypothèse très répandue) : en théorie, elle peut prendre n’importe quelle forme. La question à laquelle on a tenté de répondre était donc la suivante : si on veut mesurer la variance phénotypique produite par cette ligne noire (et la variation de cette ligne noire entre différents génotypes) dans le cas le plus général, comment faire ?
En réalité, la question, pourtant très simple à formuler, n’est pas si facile à régler. On a eu beaucoup de réflexions sur les concepts et rencontré pas mal d’obstacles, imbriqués les uns dans les autres. Lorsque nous avons abordé le sujet pour la première fois, nous (Luis-Miguel et moi) ne nous attendions pas, non seulement à tomber dans le terrier de lapin, mais en fait à creuser toute une galerie assez grande pour accueillir une ville entière de lapins !
C’est la raison principale pour laquelle il nous a fallu autant de temps pour terminer ce projet et publier l’article (ça, et aussi le fait qu’il s’agissait d’un projet secondaire pour nous deux, ce qui n’a pas aidé). Pour chaque solution trouvée, on a découvert à chaque fois de nouvelles couches de complexité à explorer… et on a dû prendre le temps d’en discuter encore et encore jusqu’à se mettre enfin d’accord (ou accepter nos désaccords, parfois) ! Ce processus dialectique s’est finalement avéré très fructueux, stimulant et riche en révélations et découvertes en tout genre.
Comment ça marche ?
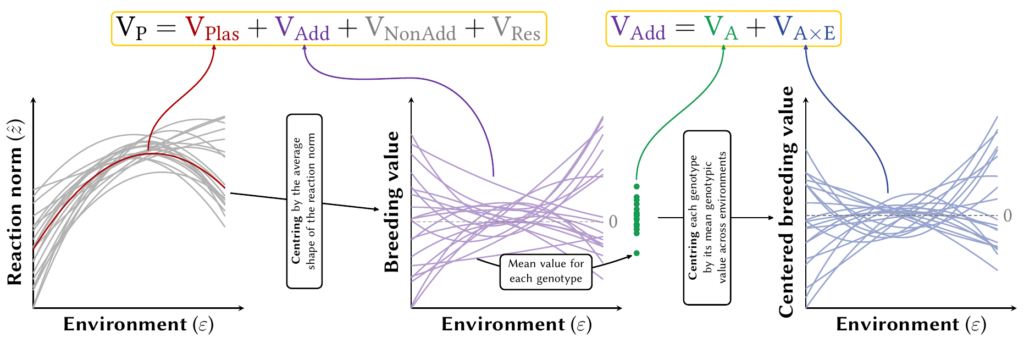
La première étape de notre cadre consiste à décomposer la variance phénotypique totale en trois composantes :
- La variance résultant de la forme moyenne de la norme de réaction à travers les différents génotypes, que nous avons noté VPlas ;
- La variance génétique résultant de la variation entre les génotypes au-delà de cette forme moyenne, que nous avons noté VGen ;
- Une variance résiduelle, pour la variation du trait phénotypique qui n’est pas prédite par la norme de réaction, que nous avons noté VRes.
En tant qu’évolutionnistes, on est intéressé par la réponse à la sélection, donc on s’est concentré sur la partie de la variance génétique VGen qui est héritable (variance génétique additive), que nous avons appelée VAdd. Et ensuite, on a décomposé davantage (je parlais de couches…) cette variance héritable totale VAdd en une variance héritable « indépendante de l’environnement » (qui serait calculée en ignorant la plasticité phénotypique), VA, d’une part ; et la variance héritable résultant de la plasticité phénotypique, que nous avons appelée VAxE, d’autre part. En divisant ces quantités par la variance totale, on peut calculer la proportion de la variance due à VPlas (P²RN) et les héritabilités correspondantes pour les composantes héréditaires (h²RN, h² et h²I).
Mais les couches de complexité ne s’arrêtent pas là : on peut également déterminer dans quelle mesure ces variations dépendent de la forme ou des paramètres des normes de réaction. Cela vaut aussi bien pour VPlas que pour les composantes héréditaires (VAdd, VA, VAxE).
En ce qui concerne la variance due à la forme moyenne de la norme de réaction (VPlas), on peut la décomposer en des variances provenant de la pente ou de la courbure moyenne de la norme de réaction sous certaines hypothèses (par exemple, la normalité). On l’a appelé la décomposition π. Les composantes héritables (VAdd, VA, VAxE) sont, par définition, additives (pour des raisons que je ne vais pas aborder ici). Ça signifie que leurs calculs sont, essentiellement, des grosses sommes de produits (une combinaison linéaire) qui dépendent de l’environnement et des paramètres de la norme de réaction. Chacun des produits (des éléments de la somme) peut être relié à un paramètre, ou un couple de paramètres, de la norme de réaction. Ça veut dire qu’on peut évaluer dans quelle mesure chacune de ces composantes héritables dépend de la variation génétique des paramètres de la norme de réaction. On a appelé ça la décomposition γ pour VAdd et la décomposition ι pour VAxE.
À quoi ça sert ?
Avec ce cadre conceptuel, on fournit plusieurs choses :
- On pense que ça clarifie certains aspects de la relation entre les normes de réaction et la variance phénotypique. Certains éléments étaient déjà connus, bien sûr, mais n’avaient pas été présentés exactement comme nous l’avons fait. D’autres encore sont, selon nous, assez nouveaux, du moins présentés avec une telle généralité.
- Ça permet de normaliser la contribution de la forme des normes de réaction à la variance phénotypique, ce qui (rappelons-le !) était notre objectif principal.
- Ça fournit un cadre très général et théoriquement fondé pour étudier la génétique quantitative des normes de réaction, et en particulier pour explorer où se situe le potentiel adaptatif héritable dans celles-ci (dans la variation du trait indépendante de l’environnement ? Dans la plasticité ? Et quel paramètre de la norme de réaction y contribue le plus ?).
- On a tout conçu de manière à ce que tout ce qui précède puisse être appliqué de la manière la plus générale possible, s’appliquant à toutes les différentes façons disponibles pour traiter les normes de réaction (à l’aide de courbes linéaires ou non linéaires, ou d’une approche discrétisée appelée « character-state »).
Pour favoriser l’adoption de cette décomposition de variance, on fournit tout ça comme une série de fonctions empaquetés dans la libraire R Reacnorm (voir aussi le dépôt Github si besoin).
Où est-ce que c’est publié ?
Puisque Luis-Miguel et moi-même occupons tous deux des postes permanents (et donc n’avons plus besoin de collection à fond les noms prestigieux de revues dans notre CV), on a décidé de soutenir l’initiative Peer Community In avec cette publication. Cette initiative vise à contourner l’économie hautement dysfonctionnelle de la publication scientifique en promouvant un processus de recommandation par les pairs basé sur des prépublications, suivi éventuellement d’une publication dans la revue en ligne Peer Community Journal, où notre article est désormais publié.
Je dois dire que le processus de publication a été super satisfaisant, avec beaucoup moins de frictions, tant pendant la phase de révision qu’après celle-ci. La révision a bien sûr été aussi stricte et exigeante que pour n’importe quelle revue sérieuse (l’historique de révision est d’ailleurs transparent et disponible en ligne si vous souhaitez le consulter). Nous avons eu beaucoup de contrôle sur l’édition de la version finale de l’article, grâce à un dialogue cordial avec l’équipe éditoriale, ce qui a été un changement très rafraîchissant par rapport à de nombreux autres journaux ! 5/5 ⭐️⭐️⭐️⭐️⭐️, recommande !
Ceci dit, ça signifie que notre papier risque de ne pas toucher une partie de son lectorat, pas encore habituer à surveiller ce secteur de la publication scientifique. Donc si vous appréciez son contenu, aidez-nous à le faire connaître autour de vous ! 🙏